
" " "
TL;DR : UNet은 contracting path에서는 context를 캡쳐하고, 대칭적인 구조를 이루는 expanding path에서는 정교한 localization을 가능하게 하는 구조이며, Data Augmentation 기법으로 Elastic deformation을 활용하여 성능 UP!.
" " "
U-Net 논문의 핵심내용
- Convolution Encoder에 해당하는 Contracting Path(내려가는부분) + Convolution Decoder(올라가는부분)에 해당하는 Expanding Path의 구조로 구성. (해당 구조는 Fully Convolution + Deconvolution 구조의 조합)
- Expanding Path(올라가는부분)에서 Upsampling 할 때, 좀 더 정확한 Localization을 하기 위해서 Contracting Path(내려가는부분)의 Feature map를 Copy and Crop하여 Concat 하는 구조.
- Data Augmentation = Random Elastic Deformation
Related Works
기존 Segmentation 접근법은 느리다!!!(왜냐? Sliding Window 패치 중복연산)
기존 Segmentation 접근법은 정확도가 떨어진다!!!(큰 사이즈 patchs는 max-pooling 많이 해야함->localization 성능 저하)
Introduction
UNet 특징
- Constracting path(내려가는부분)에서 풀링 전에 Feature map을 뽑아내서 올라가는부분(Upsampling) 시 결합을 통해해상도가 높은 수준을 유지하는 기능이 있다.
- 내려갈때 64채널 -> 맨 하단 1024채널 -> 맨위 64채널
- 각 레이어(layer;파란부분)에서 컨볼루젼연산(convolution)만 사용하며, Fully-connected layer 는 사용하지 않음.
- Overlab-tile 기법 사용(Fig. 2)

Overlab-tile
Fig. 2를 보면 오른쪽 그림에 파란색 경계상자에 흰색으로 채워지지 않은 부분이 존재한다. 이제 왼쪽 그림을 보면 채워져있지 않던 흰색 부분이 마치 데칼코마니 한것처럼 경계를 기준으로 미러링(mirroring) 되어있다.(아래그림참조)
Architecture

Contracting Path(내려가는 부분)
- 2번의 3x3 Convolution 수행
- ReLU
- 2x2 max-pooling(stride 2)
- Feature 채널 x 2
Expanding Path(올라가는 부분)
- 2x2 Convolution 수행(Up-conv 2x2)
- Feature 채널 / 2(halve; 절반으로 감소)
- 2번의 3x3 Convolution 수행
- ReLU
- Final-Layer : 1x1 convolution을 사용하여 2개 클래스를 분류
Training
컨볼루젼 연산 때 패딩(padding)을 사용하지 않기 때문에 Input size > output size.
Momentum : 0.99로 다소 큰 편 = 이전 값 더 많이 반영 가능해서 학습원활히 함.
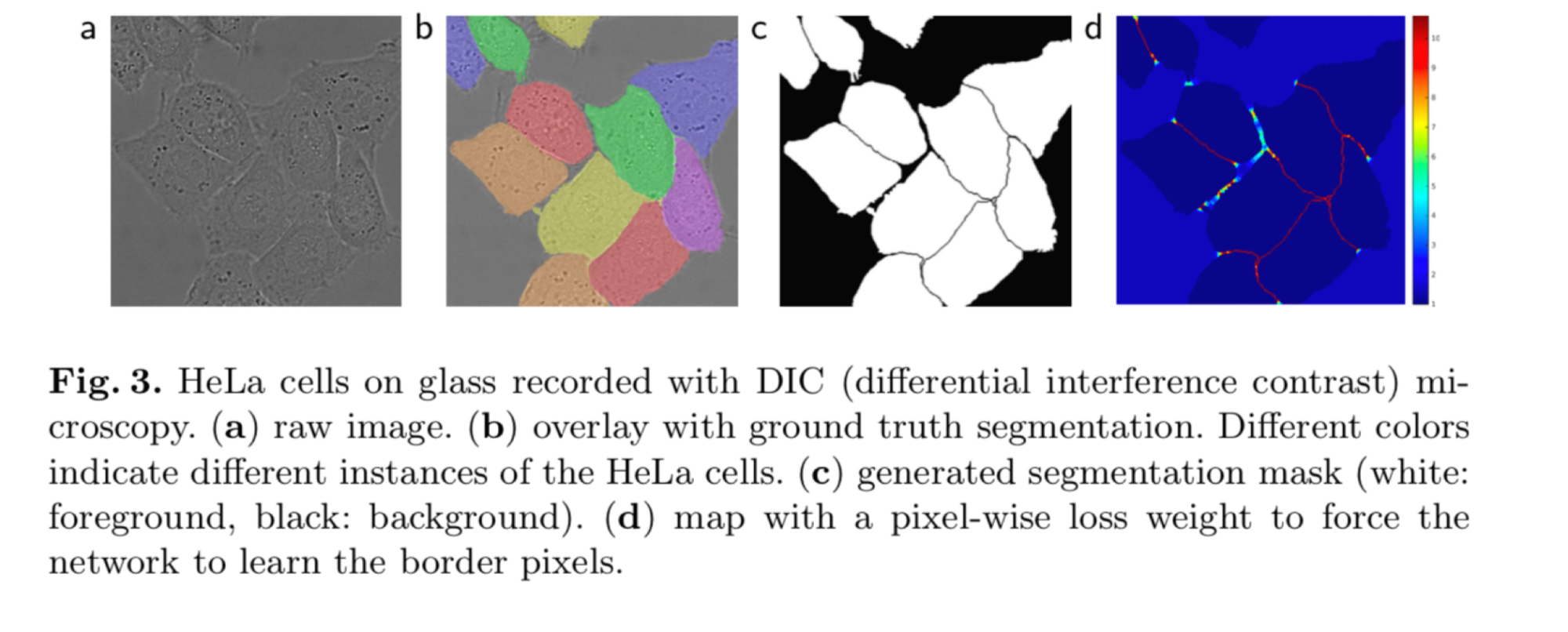
- 사진 a : raw-img(현미경사진)
- 사진 b : GT(ground-truth) 정답 라벨
- 사진 c : 배경/인스턴스 구분
- 사진 d : 세포의 경계선 학습(pixel-wise로 경계확률 높으면 빨강, 낮으면 파랑)
Softmax, Cross-Entropy loss 에 관한 내용의 설명은 다른 포스팅에서 참고했음.
내가 이해한 바로는, 각 픽셀에 가중치를 부여하는데, 어떻게 부여하느냐! 클래스의 빈도수에 따라 값을 정한다.
학습할때 데이터에 x픽셀이 배경(background)일 확률이 높은지, instance(foreground)일 확률이 많은지 빈도를 따져서 결정된다고 이해하였다.
d1 은 가장 가까운 세포까지의 거리를 나타내고, d2는 두번째로 가까운 세포까지 거리를 나타낸다.
다시말해, x는 세포들 사이에 경계를 의미하고 두 세포 사이 거리/간격이 좁으면 큰 weight를 갖고, 거리/간격이 넓으면 weight값이 낮다. 논문에서는 경계(border)의 weight map을 계산하기 위해 이미지 처리기법인 morphological operation을 적용했다고 한다.
Softmax 수식
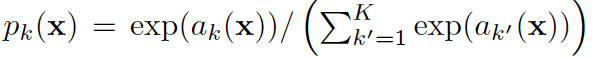
Cross Entropy Loss with w(x) 수식
각각 정답 픽셀에대한 cross entropy loss에는 w(x)라는 가중치 값이 추가되고, 여기서 의 는 정답 클래스 즉 위 softmax 수식에서 정답의 레이블에 해당하는 값을 반환하는 함수이다.
cross entropy 함수는 정답의 추정값을 log에 사용하기 때문에 이에 해당하는 정답의 확률을 가져오는 것. 수식 (1)은 loss 값에 가중치 w(x)를 곱한 형태이며 이제 우리가 살펴볼 것은 이다.

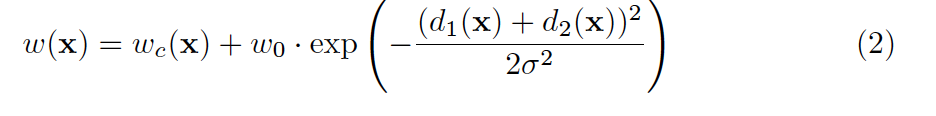
Data Augmentation
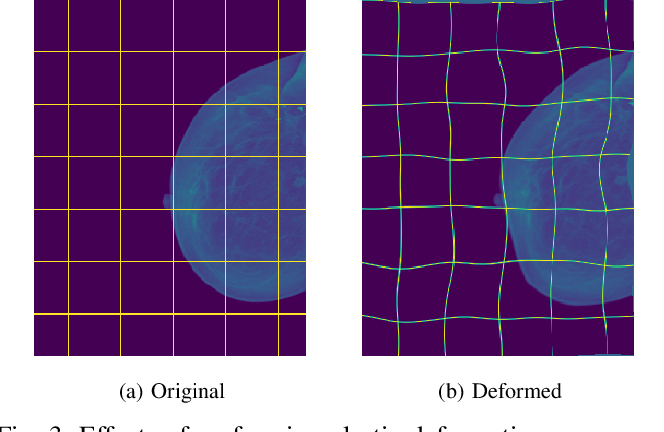
논문에서 핵심기법?으로 소개하는 것으로 elastic deformation을 통해 데이터를 확장하여 robust한 모델을 학습시킬 수 있었다고 한다. 아래 그림은 이해를 돕기 위해 가져온 것으로, elastic(유연)하게 이미지의 경계를 변형시켜 학습하여 흔들림, 왜곡된 이미지도 잘 구분할 수 있게 한다. (유연한 지수는 가우시안 분포 10픽셀 SD로 설정)
Experiments
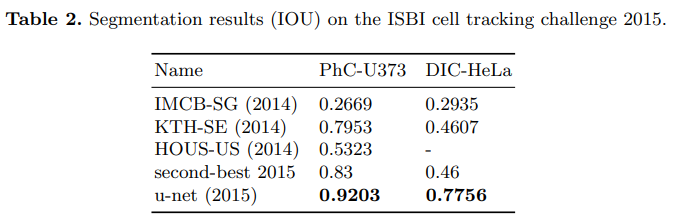
보통 Segmentation task는 IOU(Intersection over Union)기법을 통해 성능을 측정한다. 쉽게 말해 라벨영역과 예측영역이 얼마나 겹쳐있는지 계산하는 기법으로 알고있다. UNet 은 여러 Challenge에서 가장 좋은 성능을 보였다. 아래 참고.




댓글